x <- c(65, 75, 67, 79, 81, 91) # observed data
y <- 50:99
h <- 5.5
n <- length(y)
B <- numeric(n)
K <- numeric(n)Probability density function
확률밀도함수(pdf)는 확률변수의 분포를 나타내는 함수로 보통 확률변수가 연속형일 때를 지칭한다. 확률 밀도 함수는 두 가지 조건을 만족해야 한다.
모든 실수값 x에 대해
pdf 조건에서 알 수 있듯이 확률밀도함수는 확률이 아니며, 확률밀도함수를 적분해야만 확률이 나온다.
Probability density
확률 밀도는
연속형 확률 변수
만약
따라서
다시 처음 확률밀도함수의 정의에서 출발하면 확률 변수
이 정의도 문제가 있는데 정의 그대로
Density estimation 의미
통계학에서 density estimation은 보통 분포(확률밀도함수)의 형태를 가정하고 데이터를 이용해서 모수를 추정하는 방식이다.(parametric density estimation이라고도 한다)
반면에 kernel density estimation에서는 분포의 형태를 가정하지 않고 관찰된 데이터만을 이용해서 분포를 추정한다. (non-parametric density estimation이라고도 한다)
Kernel density 정의
간격을 좁게 하면 undersmooth되고, 반면에 간격을 넓게 하면 oversmooth된다.
kernel function 종류

kernel의 형태는 정의하기에 따라 여러 종류가 있다. kernel 함수의 형태에 따라 추정되는 밀도의 형태가 조금씩 바뀐다. 다만 전반적인 추정된 밀도의 형태는 거의 유사하다(MISE를 기준으로 kernel별 점근적 상대 효율성을 비교해보면 Epanechnikov kernel이 1로 가장 좋지만 다른 kernel function과 큰 차이를 보이지 않는다).
kernel function을 정하는 것 보다는 bin width를 정하는 것이 더 중요하다. rectangular kernel일 경우 histogram density estimation과 같다.
Gaussian kernel example
for (j in 1:n) {
A <- 1/(h*sqrt(2*pi))
B[j] <- (-0.5)*((y[j] - 65)/h)^2
K[j] <- A*exp(B[j])
}
plot(y, K, type = 'l', main = 'kernel at xi = 65')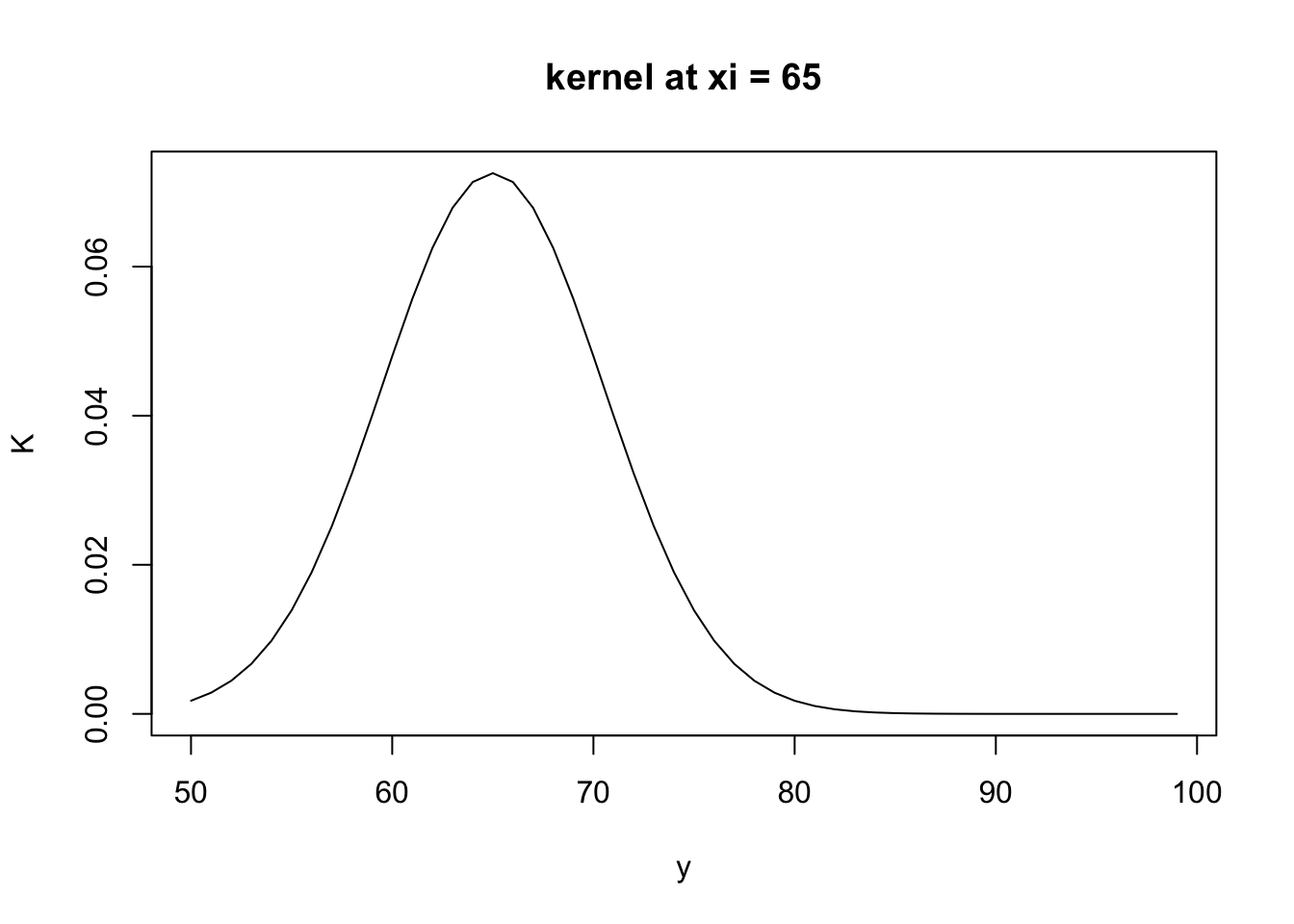
각
m <- length(x)
B <- matrix(0, nrow = n, ncol = m)
K <- matrix(0, nrow = n, ncol = m)
for (i in 1:m) {
for (j in 1:n) {
A <- 1/(h*sqrt(2*pi)*m)
B[j, i] <- (-0.5)*((y[j] - x[i])/h)^2
K[j, i] <- A*exp(B[j, i])
}
}
plot(y, K[,1], type = 'l', main = '', xlim = c(45, 110), ylim = c(0, 0.04))
for (i in 2:6) {
lines(y, K[,i], type = 'l', main = '')
}
# 최종 kernel
K <- round(K, digit = 7)
d <- rowSums(K)
lines(y, d, type = 'l', main = 'Kernel density')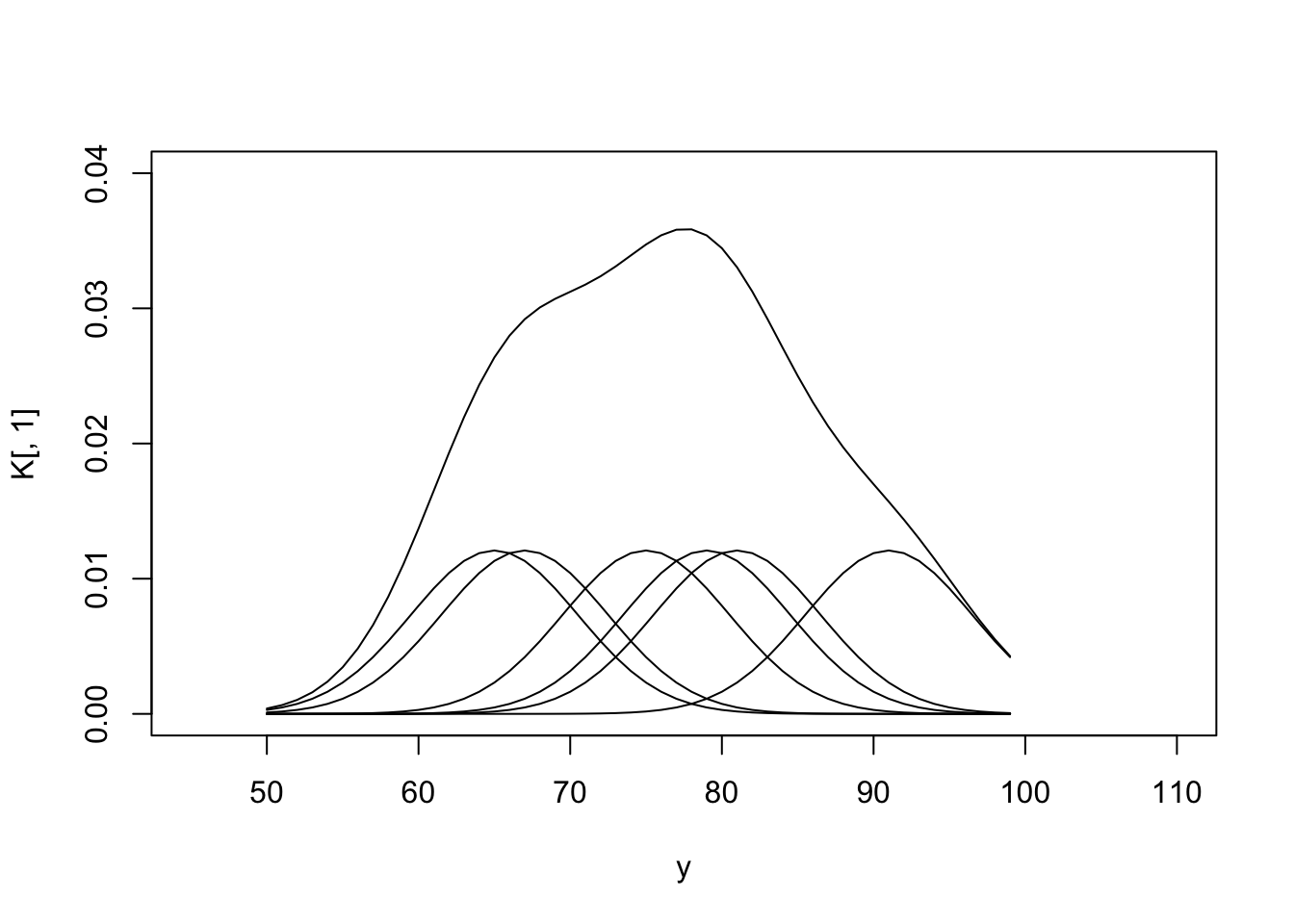
x <- c(-0.77, -0.6, -0.25, 0.14, 0.45, 0.64, 0.65, 1.19, 1.71, 1.74)
y <- seq(-4, 4, 0.1)
n <- NROW(x)
m <- NROW(y)
B <- matrix(0, nrow = m, ncol = n)
K <- matrix(0, nrow = m, ncol = n)
h <- 0.25
for (i in 1:n) {
for (j in 1:m) {
A <- 1/(h*sqrt(2*pi)*n)
B[j, i] <- (-1/2)*((y[j] - x[i])/h)^2
K[j, i] <- A*exp(B[j, i])
}
}
plot(y, K[,1], type = 'l', main = 'h = 0.25', ylim = c(0, 0.55), ylab = '', xlab = '')
for (i in 2:10) {
lines(y, K[,i], type = 'l', main = '')
}
K <- round(K, digit = 7)
d <- rowSums(K)
lines(y, d, type = 'l', main = '')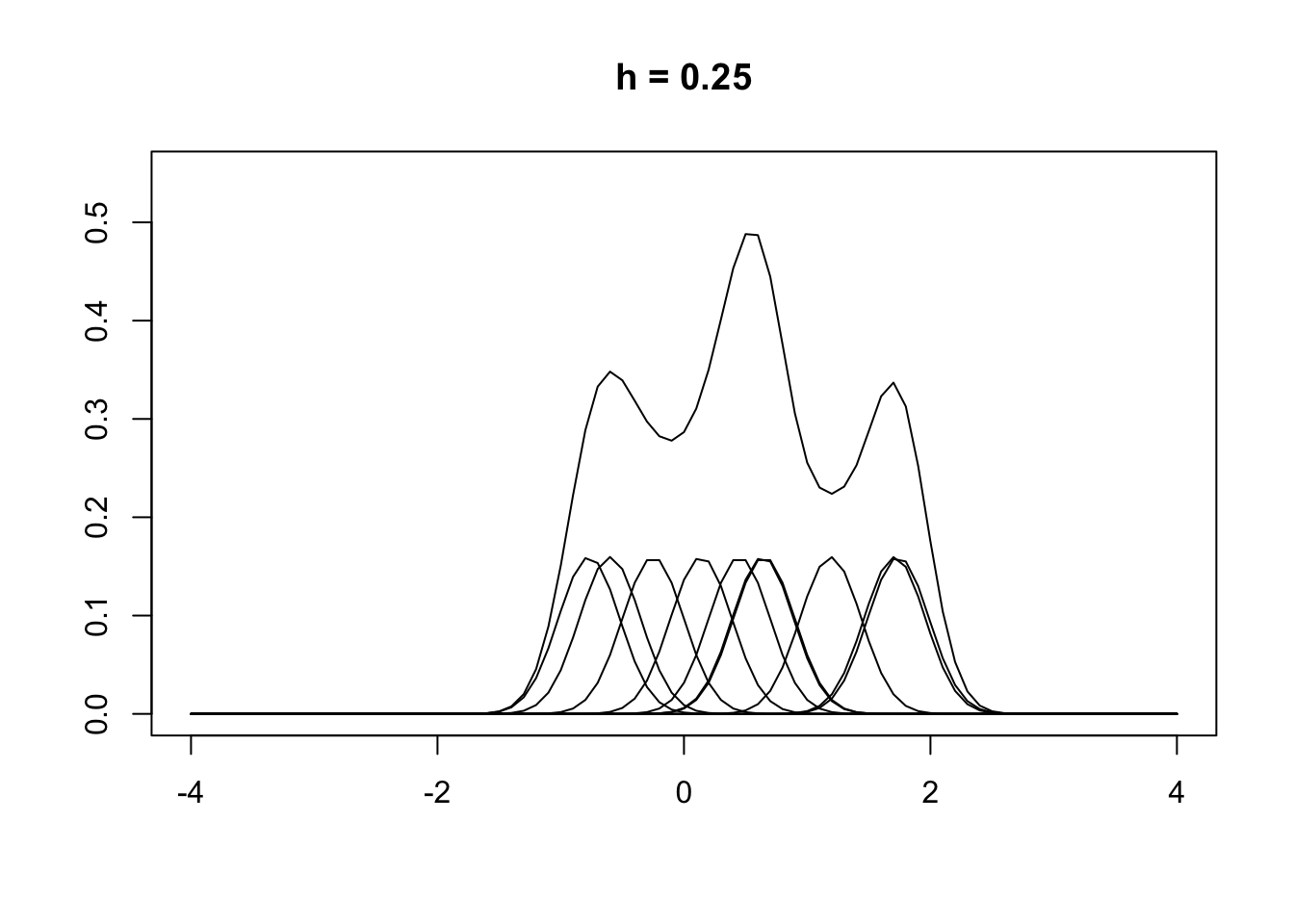
x <- c(-0.77, -0.6, -0.25, 0.14, 0.45, 0.64, 0.65, 1.19, 1.71, 1.74)
y <- seq(-4, 4, 0.1)
n <- NROW(x)
m <- NROW(y)
B <- matrix(0, nrow = m, ncol = n)
K <- matrix(0, nrow = m, ncol = n)
h <- 1
for (i in 1:n) {
for (j in 1:m) {
A <- 1/(h*sqrt(2*pi)*n)
B[j, i] <- (-1/2)*((y[j] - x[i])/h)^2
K[j, i] <- A*exp(B[j, i])
}
}
plot(y, K[,1], type = 'l', main = 'h = 1', ylim = c(0, 0.55), ylab = '', xlab = '')
for (i in 2:10) {
lines(y, K[,i], type = 'l', main = '')
}
K <- round(K, digit = 7)
d <- rowSums(K)
lines(y, d, type = 'l', main = '')
Boundary kernel
원점에서 불연속점이 존재하거나
set.seed(1)
x <- rexp(1000, 1)
plot(density(x), xlim = c(-1, 6), ylim = c(0, 1), main = '')
abline(v = 0)
y <- seq(0.001, 6, 0.01)
lines(y, dexp(y, 1), lty = 2)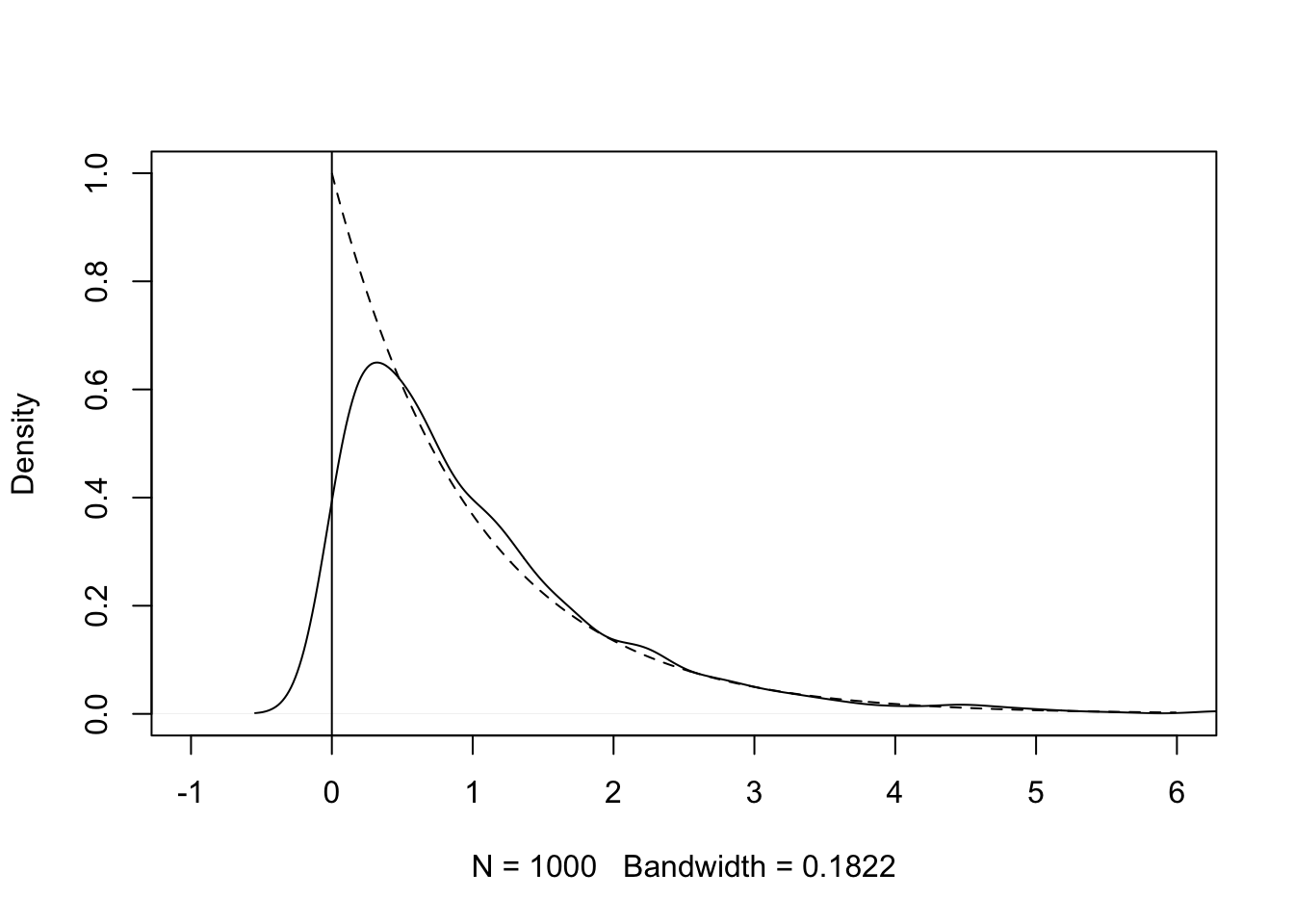
Reflection boundary technique
전체 데이터
즉, 대칭인 2개의 데이터셋에 대해서 각각 KDE를 구한다. 두 개의 KDE를 합한 KDE를 구하고 원 데이터 범위의 KDE만 남긴다.
set.seed(1)
x <- rexp(1000, 1)
z=-x
plot(density(x), xlim = c(-6, 6), ylim = c(0, 1), main = '', col = 'red')
abline(v = 0)
y <- seq(0.001, 6, 0.01)
lines(density(z), xlim = c(-6, 6), ylim = c(0, 1), main = '', col = 'blue')
xx <- c(x, -x)
g <- density(xx, bw = bw.nrd0(x))
a <- seq(-6, 6, 0.01)
ghat <- approx(g$x, g$y, xout = a) # 지정된 수의 근사 함수 값을 반환
fhat <- 2*ghat$y
bw <- paste('Bandwidth = ', round(g$bw, 5))
lines(a, fhat, type = 'l', xlim = c(-6, 6), ylim = c(0, 1), main = '', xlab = bw, ylab = 'density')
abline(v=0)
y <- seq(0.001, 6, 0.01)
lines(y, dexp(y, 1), lty = 2)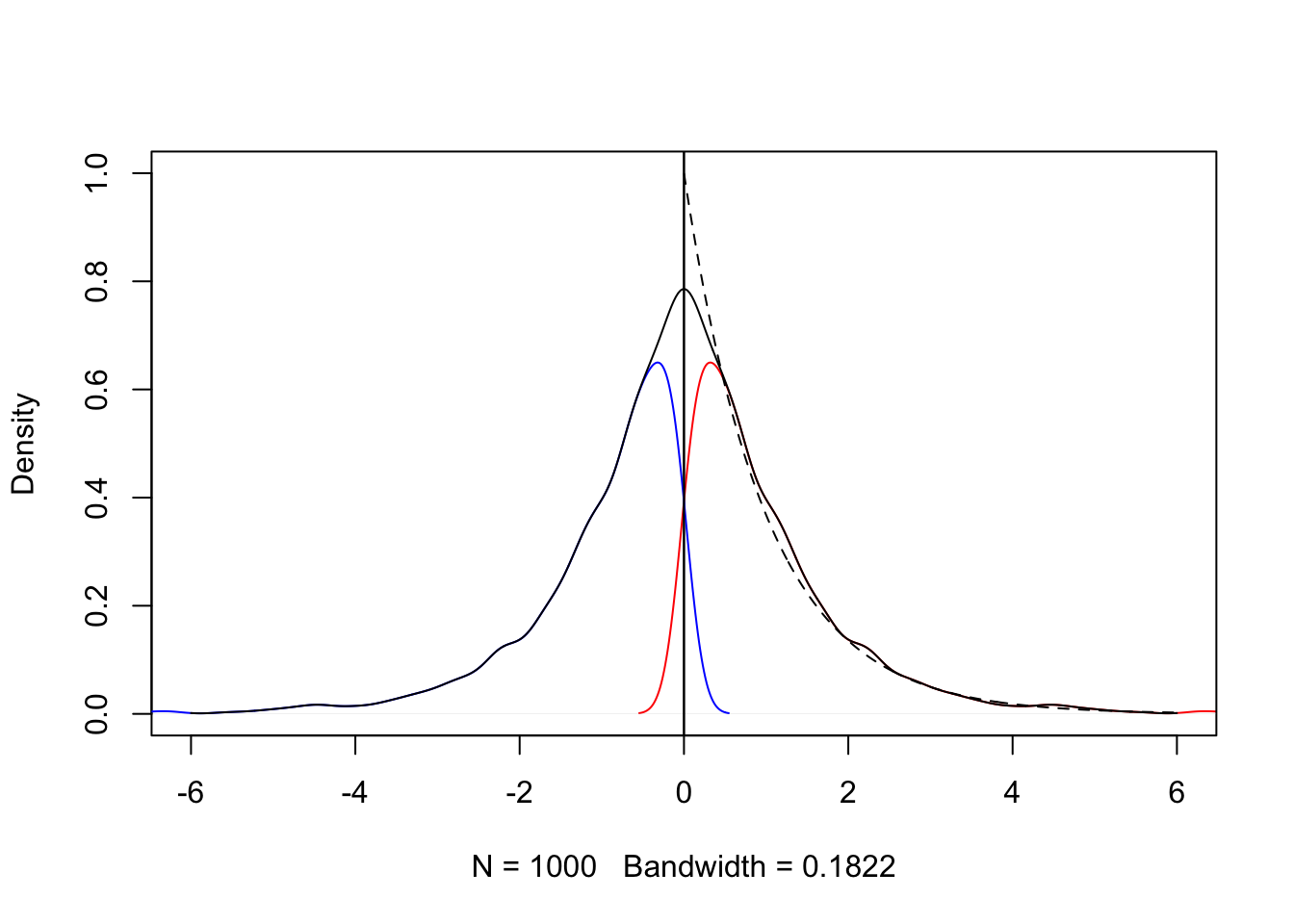
참고 2 : statistical computing with R second edition
Citation
@online{don2021,
author = {Don, Don and Don, Don},
title = {Kernel Density Estimation},
date = {2021-06-22},
url = {https://dondonkim.netlify.app/posts/2021-06-12-kernel-density-estimation/KDE.html},
langid = {en}
}